生物信息学
3.6 宏基因组分析专题
桂松涛
songtaogui@163.com

什么是宏基因组
|
|

|
为什么研究宏基因组

为什么研究宏基因组

如何研究宏基因组
|

|
如何研究宏基因组
测序和分析步骤概览

16s rRNA 测序

16s rRNA 测序
16s rRNA 测序: 分析步骤

质量控制
Fastq 格式

质量控制
Fastq 质量值
|
|
|
|
质量控制
ASCII码表

质量控制
Fastq 质量质控: 测序reads各位置质量值分布
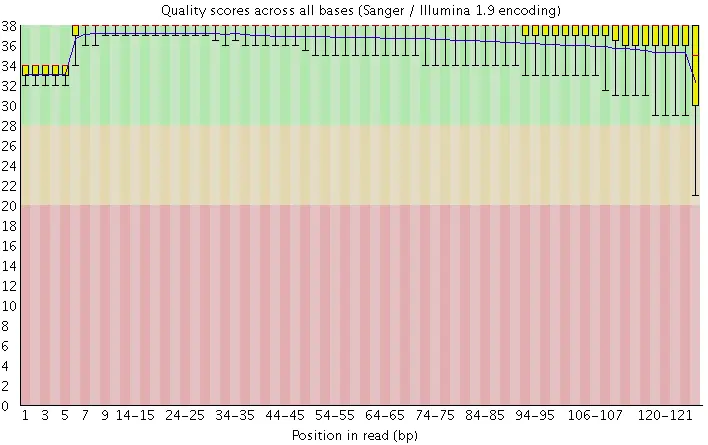
质量控制
Fastq 质量质控: 测序reads各位置质量值分布

质量控制
Fastq 质量质控: 碱基总体质量值分布

质量控制
Fastq 质量质控: Reads各位置上碱基比例分布
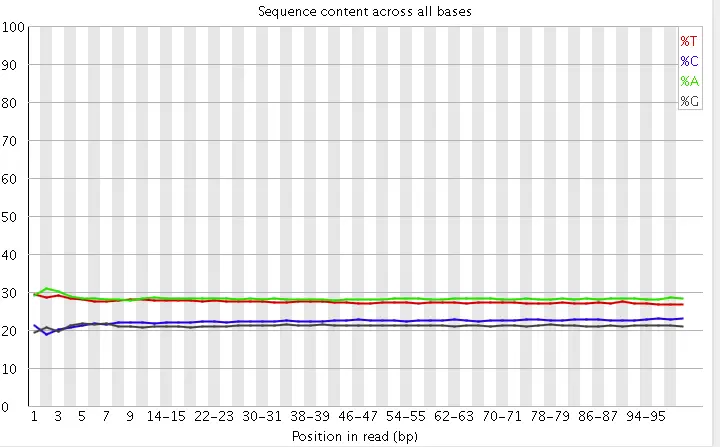
质量控制
Fastq 质量质控: GC含量分布
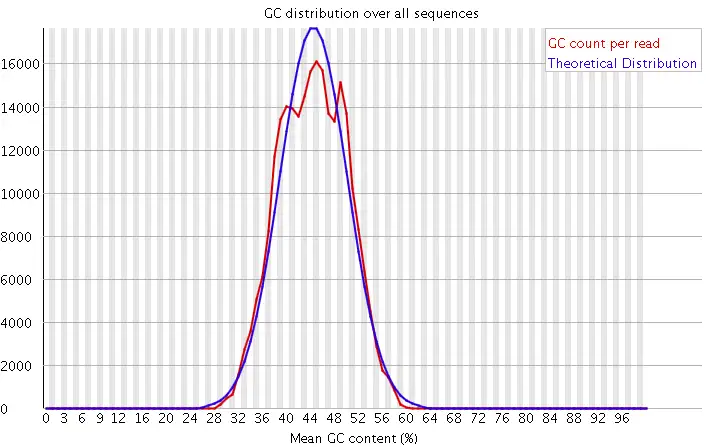
质量控制
Barcode和Demultiplex
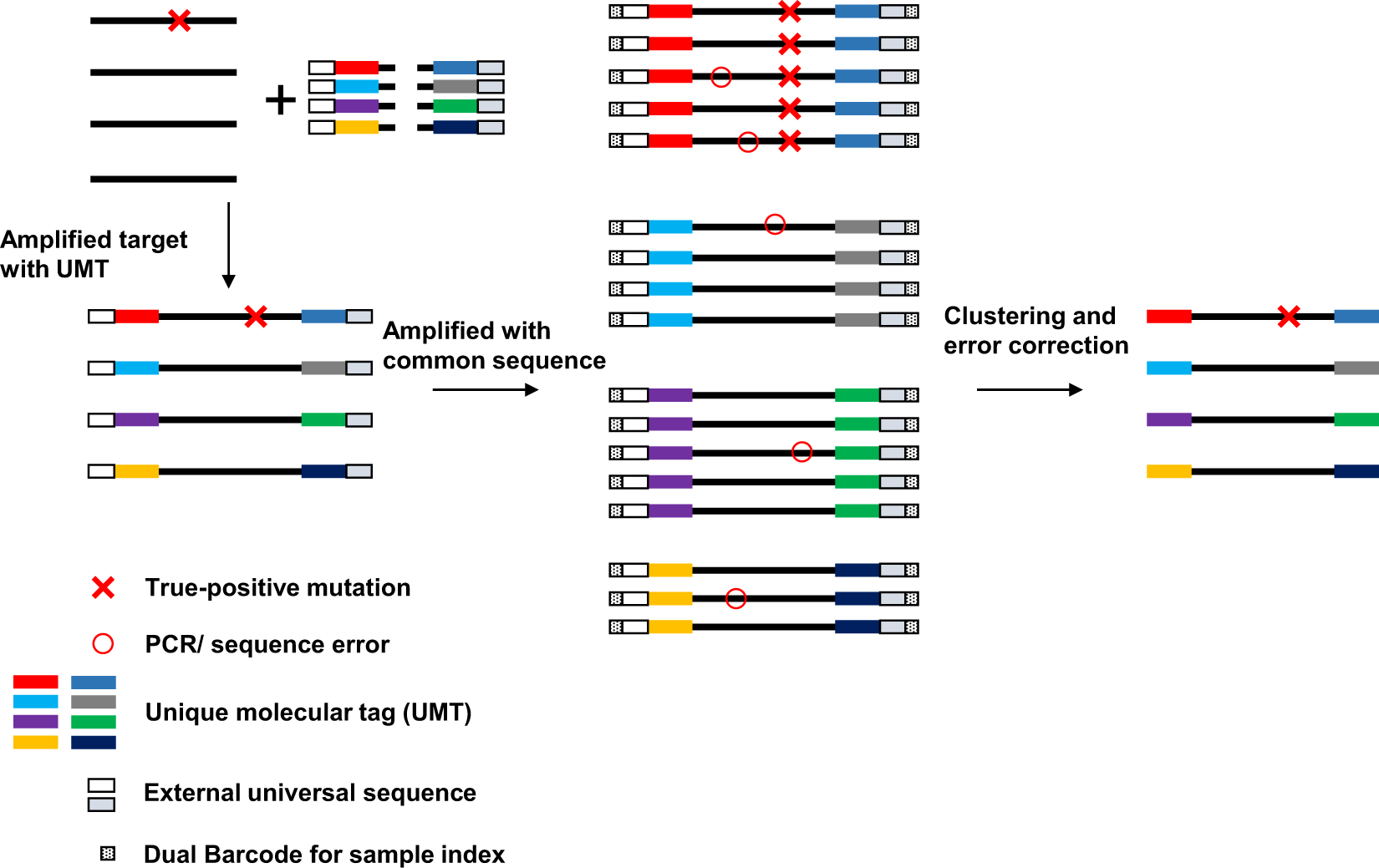
质量控制
检测和去除嵌合体(Chimera)

OTU鉴定
OTU聚类方法
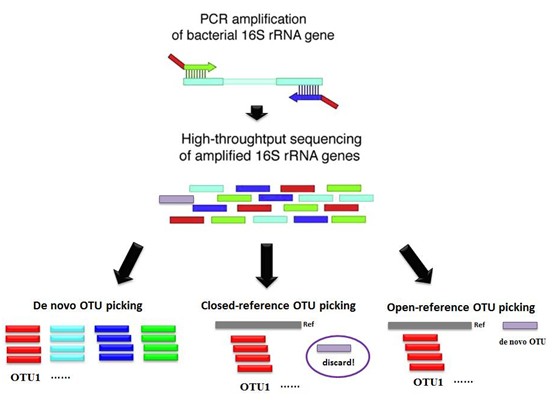
OTU聚类方法
常用OTU聚类数据库

GreenGene和RDP数据库更新太慢,一般采用Silva数据库进行分析