生物信息学
关于本课程的一些说明
桂松涛 Blog
songtaogui@163.com

2023年10月
WHY? 事要知其所以然
生物信息工具的使用也需要理解背后的原理
|
PCR:
|
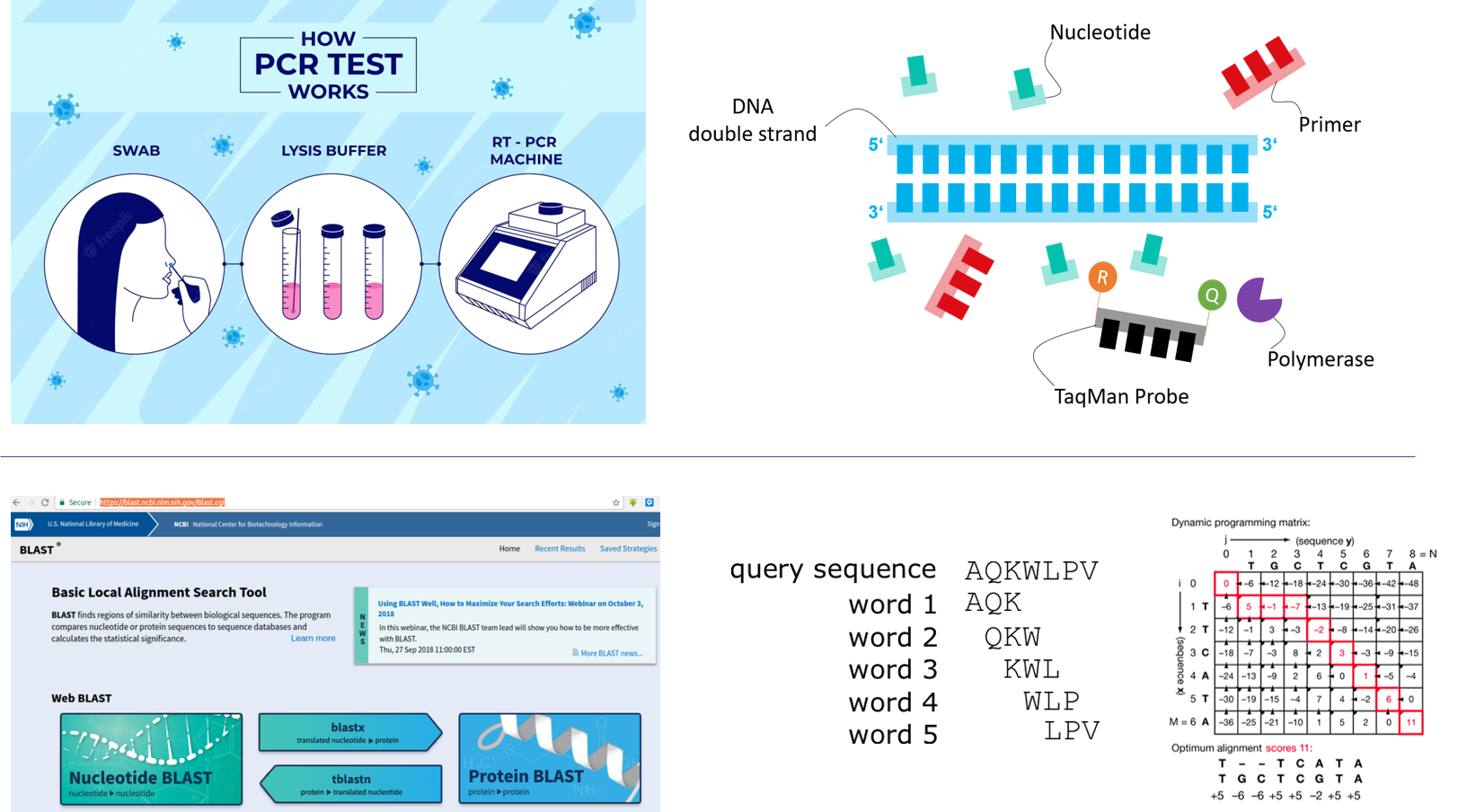
|
生信专业从业者的技能树
|
Bioinformatics ≈ Biology + Programming + Mathematics 延伸阅读: 生物信息等级划分 |
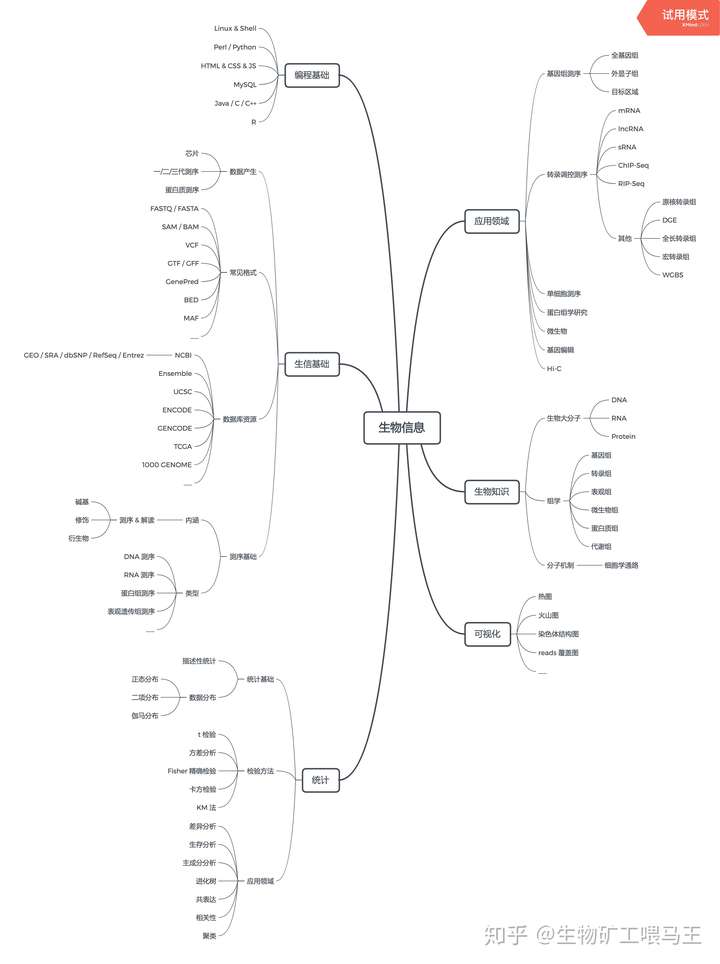
|
生信工具使用者的加点建议
课程概览
|
|
绝知须躬行
课上内容以框架、大纲为主;
提供自学资源、资料;
90%以上的内容需要课下自学掌握;